- HOME
- 製品・サービス
- 目的別:アプリケーション選択ガイド
- CRISPRスクリーニング解析
CRISPRスクリーニング解析

解析概要
CRISPR gRNAライブラリを使用したゲノムワイドスクリーニングにご利用いただける次世代シーケンス解析サービスです。gRNA領域のPCR産物をご提供いただき、ディープシーケンス、gRNAのリードカウントを行います。

サービス内容

データ量について
データ量は、リードペア数 x リード長 x 2(ペア)となります。150bpで1,000万リードペアを読む場合、10,000,000 x 150bp x 2 = 3,000,000,000 b = 3Gb となります。
また、リードペア数 / gRNAの種類数 = カバレッジ となります。CRISPRスクリーニング解析には、一般的に x100 ~ 500 カバレッジ程度の深度が推奨されています。
例えば、12万種類のgRNAライブラリについて平均100カバレッジとなるデータを取得するには、12万 x 100 = 1,200万 リードペアとなり、データ量は 12,000,000 x 150bp x 2 = 3.6Gbのように計算します。
ご使用のgRNAライブラリの種類数、および、ご希望のカバレッジに応じて取得データ量(リードペア数)を ご設定ください。
納品データ
| 品名 | 内容 | 形式 | 詳細 |
| 書面 |
・作業報告書
|
- | 作業内容の概要 |
|
・Sequencing Report
|
- | シーケンスデータに関する解説書 | |
| 記録メディア ・DVD-R ・USBメモリ ・HDD 等 |
・シーケンスデータ
|
fastq / txt / xlsx | シーケンスで得られた配列データと対応するクオリティスコア(fastq)、および、シーケンスのサマリー情報 |
|
・PreProcessingデータ
|
fastq / txt / xlsx | データQCやトリミングなどの前処理を行ったデータ、および、前処理のサマリー情報 | |
|
・gRNAカウントデータ
|
txt / xlsx | MAGeCKプログラムを使用して算出した、各検体のgRNAのカウントデータ データ見本1 | |
|
・検体比較結果
|
txt / xlsx | MAGeCKプログラムを使用して算出した、検体間・群間の比較結果 データ見本2 / データ見本3 | |
|
・リファレンス情報
|
txt | 解析に使用した、gRNAライブラリの配列情報 | |
|
・パラメータシート
|
txt | 解析に使用したプログラムと条件値の一覧 | |
|
・Analysis Report
|
データ解析に関する解説書 | ||
|
・付加情報
|
fastq閲覧用のフリーソフトのご案内や、ライブラリ構造の図解 |
データ見本1 : gRNAのリードカウントテーブル
| gRNA ID | gRNA Sequence | SampleA Count |
| ID_0001 | AAATACAAACAACTCTGAG | 213 |
| ID_0002 | GAAGTGTCCCAGGGCGAGG | 294 |
| ID_0003 | ACTCTATCACATCACACTG | 35 |
データ見本2 : gRNAのカウント比較テーブル
| gRNA ID | Control | Test | LOG2FC | p_value | FDR | High in Treat | ||
|---|---|---|---|---|---|---|---|---|
| SampleA | SampleB | SampleC | SampleD | |||||
| ID_0001 | 213 | 274 | 883 | 175 | 1.12 | 0.9996 | 0.00699 | FALSE |
| ID_0002 | 294 | 412 | 1554 | 1891 | 2.29 | 0.0000 | 0.00000 | TRUE |
| ID_0003 | 421 | 368 | 566 | 759 | 0.75 | 0.9995 | 0.00726 | FALSE |
データ見本3 : 遺伝子のnegative/positive selection情報
| Gene | negative selection | positive selection | ||||
|---|---|---|---|---|---|---|
| p_value | FDR | Rank | p_value | FDR | Rank | |
| RPS5 | 0.000 | 0.000236 | 1 | 1.000 | 1.000 | 19147 |
| YARS | 0.000 | 0.000236 | 2 | 1.000 | 1.000 | 19146 |
| GTF2B | 0.000 | 0.000236 | 3 | 1.000 | 1.000 | 19145 |
| NRF1 | 0.000 | 0.000236 | 4 | 1.000 | 1.000 | 19144 |
| NLE1 | 0.000 | 0.000236 | 5 | 1.000 | 1.000 | 19143 |
ご用意いただくもの
サンプル
gRNA領域のPCR産物
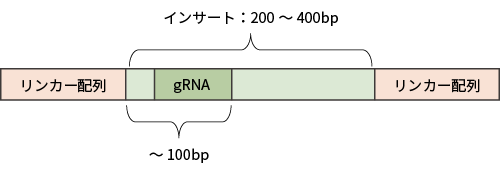
■ PCR産物の構造
-
- 1. 弊社指定のリンカー配列(※配列はお問い合わせください)を付加したプライマーでPCRを実施し、そのPCR産物(リンカー配列付き)をご提供ください。
- 2. リンカー配列を除くインサートサイズは200~400bp程度、その内5’側の100bp以内にgRNA領域を含むようプライマーを設計してください。(上図参照)
- 3. ご提供いただくPCR産物の量は5ng(0.2ng/μL)以上を目安にご用意ください。
- 4. PCR産物は精製を行い、プライマーや非特異的産物を除去してください。
1st PCRに使用する鋳型量について
CRISPRスクリーニング実験でNGS解析を実施する際に行う1st PCRでは、スクリーニング処理後の細胞内に含まれるsgRNAのバリエーションを充分にカバーするため、ゲノム抽出の際の細胞数や1st PCRに投入する鋳型の量を検討する必要があります。
ヒトやマウスの1細胞内に含まれるゲノムDNA重量は約6.6~7pgと見積もられますので、例えば約5万種類のsgRNAライブラリを対象として、全sgRNAの種類数に対し300~500カバレッジを想定する場合、鋳型の必要量は約100ug程度と見積もられます。
ご利用のsgRNAやスクリーニング条件に応じて、必要な鋳型量を算出し、PCRボリュームや反応本数を適宜調整してください。
コントロールサンプルについて
検体のgRNAリードカウントはコントロールサンプルが無くても可能ですが、スクリーニングによるゲノム編集への影響を比較検証するためには、スクリーニング前(gRNAエンリッチ前)のコントロールサンプルについても同時にデータ取得することをお勧めいたします。
コントロールサンプルは、トランスフェクション後の培養細胞のday0(未処理)サンプルや、トランスフェクション前のgRNAライブラリなどが使用されます。
参照データ
gRNAリスト
gRNAリードカウントには、gRNAライブラリの配列情報が必須となります。
購入先のURLやカタログ番号等、配列情報の入手経路をご教示いただくか、下表のようなID、配列、対象遺伝子の対応表をご提供ください。
■ gRNAリストの例
| gRNA ID | gRNA配列 | Gene |
|---|---|---|
| ID_0001 | AAATACAAACAACTCTGAG | GeneA |
| ID_0002 | GAAGTGTCCCAGGGCGAGG | GeneB |
| ID_0003 | ACTCTATCACATCACACTG | GeneC |
記入書類
注文書
下記より注文書をダウンロードいただき、必要事項をご記入ください。
Illumina注文書(CRISPRスクリーニング解析用)