マイクロアレイ解析用RNAの品質管理
高品質なRNAサンプルを確保することは、マイクロアレイ実験の成功の基盤となります。
このページでは、RNAサンプルの品質管理に関する重要なポイントや推奨される手法について解説し、信頼性の高い解析結果を得るためのガイドラインを提供します。
RNA品質がアレイデータに与える影響
マイクロアレイ解析では、RNAの純度・分解度・品質の均一性が結果の正確性と再現性に大きく影響を及ぼします。
| 純度: フェノールやクロロホルムなどの有機溶媒が混入すると、アレイ実験の酵素反応が阻害され、検体間でRNAの標識効率に差が生じる恐れがあります。 DNAが混入すると、RNA濃度が正確性に測定できなくなり、スタートの真のRNA量にばらつきが生じます。 |
|
| 分解度: 分解が進んだRNAサンプルでは、発現変動遺伝子に偽陽性が多くなる傾向があります。 |
|
| 均一性: 同一実験系のサンプルは、状態が均一であることが理想的です。 例えばRIN7~9にばらつきがあるサンプルセットより、全てRIN7前後のサンプルセットの方が同群内の再現性が良いケースが多いです。品質差は調製条件の差を反映しており、それによって意図しない発現変動が生じている可能性が高いためです。 |
RNAの抽出と保存
推奨の抽出方法
HSSでは QIAGEN社 RNeasyシリーズ での抽出・精製を推奨しております。
miRNAマイクロアレイ解析用には、miRNeasy等のmiRNAを含むTotal RNA精製用のキットをご使用ください。miRNA画分を濃縮・精製する必要はありません。
別売の RNase-Free DNase Set や RNeasy Plus Universal Mini Kit に含まれる gDNA Eliminator Solution によるDNA除去もお勧めいたします。(非常に微量のサンプル等でロスが心配される状況では、省略した方が良いケースもあります。)
抽出したRNAはRNaseフリー水に溶解してください。DEPC処理水は反応を阻害する恐れがありますので、使用しないでください。
保存方法
-80℃での保存が最も推奨されます。
サンプルチューブはRNase-freeの製品をお勧めいたします。サンプル名のラベリングでシールを利用される場合は、凍結によって剥がれてしまわないか、粘着強度をご確認ください。
何度かサンプルを使用する予定がある場合は、必要に応じて小分けにして保存することで、凍結融解によるダメージを防ぐことができます。
RNA分解の防止策
RNAは非常に不安定な分子であり、適切な取り扱いを誤ると簡単に分解してしまいます。RNAの分解を防ぐためには、RNaseZapによる器具や作業台の除染、RNase-free・ディスポーザブル製品の使用といった基本的な注意事項を配慮した上で、最も重要なのはサンプル採取~調製を極力迅速に処理することです。
採取後、下記いずれかの処理が完了するまでは直ちに行う必要があります。
これらのグアニジン塩ベースのタンパク質変性剤は、RNaseを阻害します。
充分に均一になるようホモジナイズしてください。不完全な破砕はRNA分解の原因となります。
2. 液体窒素で瞬間凍結する
抽出作業まで絶対に融解しないように注意が必要です。
3. RNAlater等の安定化試薬で処理する
安定化試薬が効果的に浸透しやすいよう、検体を5mm角程度にカットしてください。
大きすぎると安定化される前に分解が進んでしまいます。
組織の種類によって内在性RNaseの活性が異なり、中には分解のないRNAを抽出するのが難しいものも存在します(下図)。組織を切り出すサイズ感や破砕がスムーズに進行できるかも重要なポイントです。初めて取り扱う検体では、いくつかの手順を比較するパイロット実験を行った方が良い可能性もあります。
| 図1 組織によるRNase活性の高さ | 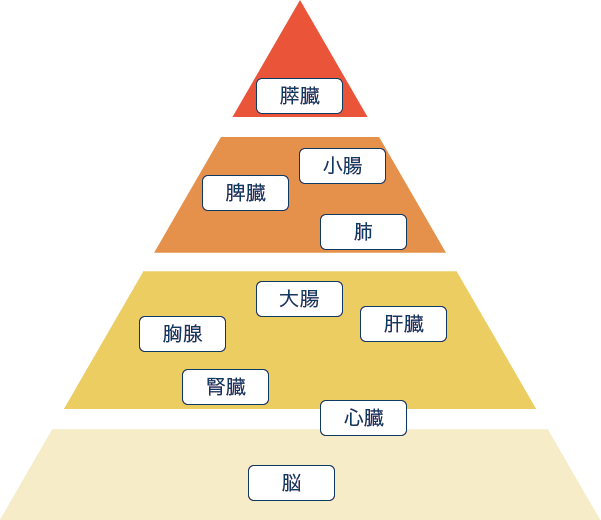 |
| 参考: Munro, J., Gillen, S. L., Mitchell, L., Laing, S., Karim, S. A., Rink, C. J., Waldron, J. A. and Bushell, M. (2023) Optimisation of sample preparation from primary mouse tissue to maintain RNA integrity for methods examining translational control. Cancers, 15(15), 3985. (doi: 10.3390/cancers15153985) |
RNA品質評価① - 濃度と純度 -
NanoDropによる吸光度測定で、RNAの濃度・純度を確認します。
濃度の測定
一般的なサンプルの場合、下記の基準を目安にサンプルをご準備いただければ安心です。
表1 マイクロアレイ解析用RNAサンプル 推奨品質基準
| サンプル量 | 500 ng 以上 |
| 濃度 | 50 ng/uL 以上 |
| 液量 | 10 uL 以上 |
| A260/A280 | 1.8 ~ 2.1 |
| A260/A230 | 2.0 以上 |
この基準は、
- – 夾雑物による見かけのRNA濃度が高く見込まれている場合
- – 品質検査やアレイ解析において、最適な濃度
- – サンプル特性や製造トラブルによって再実験を行う場合の予備量
等のリスクが考慮されているため、採取が難しい貴重なサンプルでは、基準を満たすことが難しいことがあります。そのような場合には、解析に必要な下限量(表2)を参照してください。
表2 マイクロアレイ解析に必要な最小量
| RNA品質検査(液量) | 5 uL |
| 遺伝子発現アレイ 8x60K、8x15K | 10 ng |
| 遺伝子発現アレイ 4x44K | 25 ng |
| miRNAアレイ | 100 ng |
ただし、アレイ解析にインプットする量が多い方が検出できるプローブ数も多くなりますので、極力、下限量ではなく充分量をご準備いただくことをお勧めいたします。
純度の確認
純度が表1の基準を満たしているかご確認ください。純度の高いRNAの吸収スペクトルは図2のような特徴を持ちます。230nmで吸光が低くなり、260nmで最大となります。長波長側の吸光度は0で安定しています。
| 図2 純度が高いRNAの吸収スペクトル |
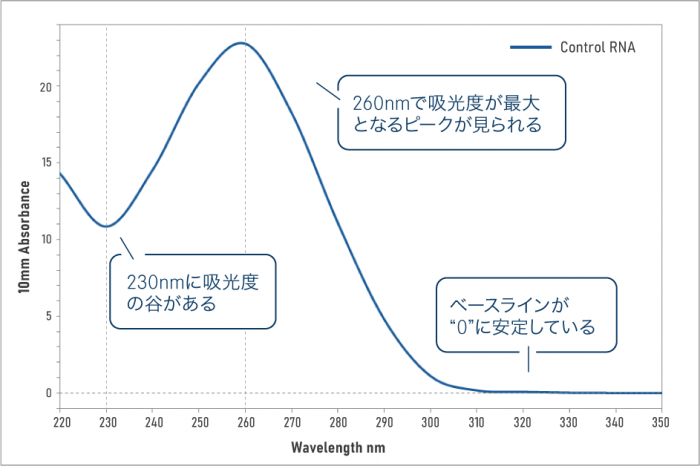 A260/A280 = 2.05 | A260/A230 = 2.10 |
一般的にRNA抽出に用いられる試薬(フェノール系溶解Buffer、グアニジンイソチオシアネートの溶解Buffer)の吸収スペクトルと、RNAに混入したときの見え方の例を示します。
| 図3 よくある混入物のスペクトルの例 | |
A フェノール系溶解Bufferの吸収スペクトル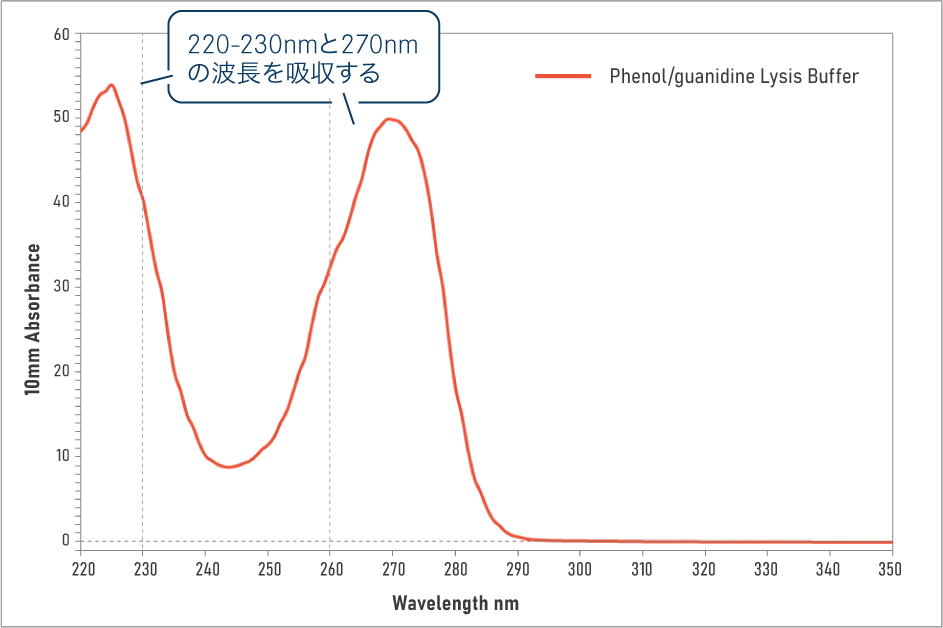 A260/A280 = 1.81 | A260/A230 = 0.81 |
B フェノールが混入したRNA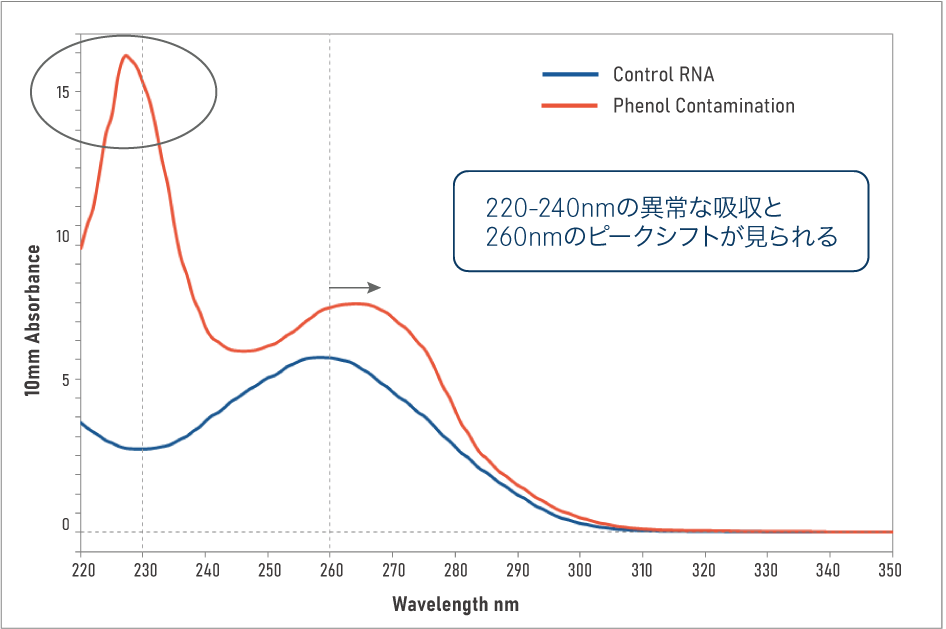 A260/A280 = 1.86 | A260/A230 = 0.50 |
C RNA抽出キットの溶解Bufferの吸収スペクトル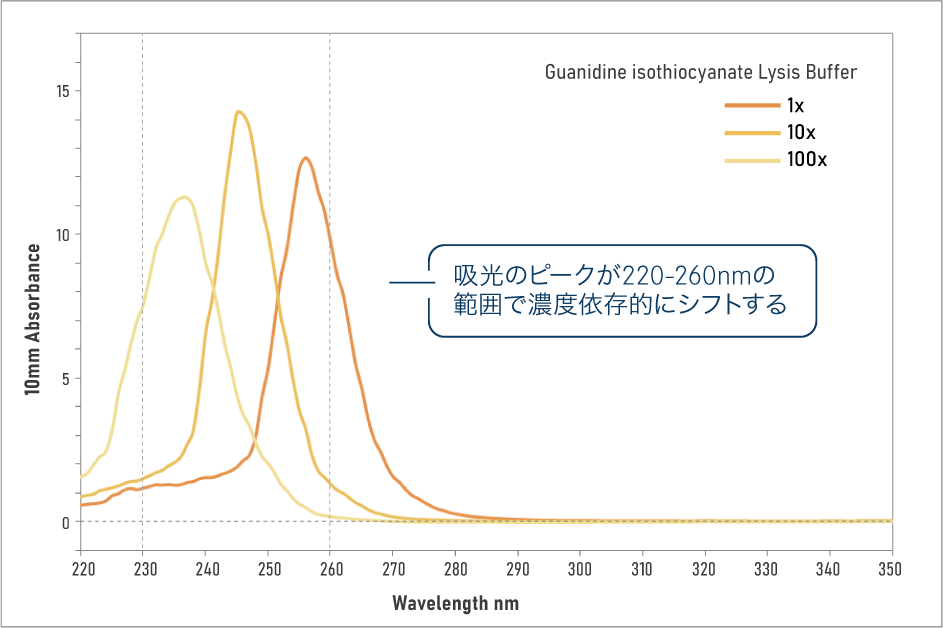 A260/A280 = 39.47 | A260/A230 = 8.46 |
D グアニジンイソチオシアネートが混入したRNA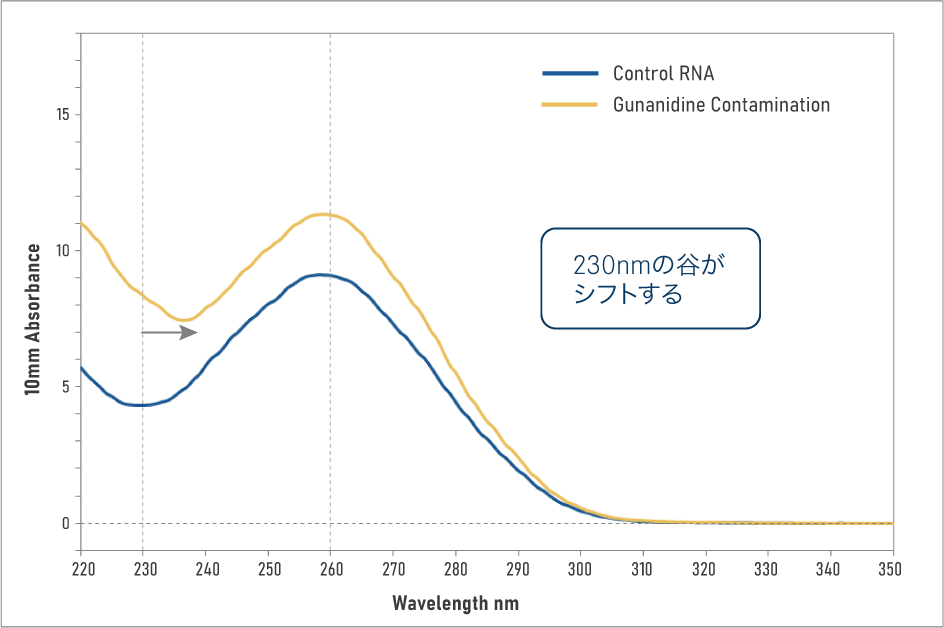 A260/A280 = 2.05 | A260/A230 = 1.36 |
フェノールやクロロホルム等の有機溶媒は酵素反応を阻害する恐れがあるため、これらの混入が疑われるサンプル(図3B)ではRNeasy等のスピンカラムを用いた精製をお勧めします。
一方で、RNeasy等のスピンカラムタイプの抽出キットで得られたRNAでは、A260/A230比が低くても(図3D)アレイ解析に影響することは経験上ほとんどありません。グアニジンイソチオシアネートによるA260/A230比の低下がダウンストリームアプリケーションに及ぼす影響についての詳細は、QIAGEN社のFAQ及びニュースレターを参考にしてください。
Qubitの利用
Qubit等の蛍光定量装置をお持ちの場合は、NanoDropとQubitの濃度測定値を比較することをお勧めします。QubitはRNAを特異的に検出するため、測定値に差がある場合は汚染物質やDNAの混入可能性を示唆します。Qubitによる測定は、特に低濃度サンプルの正確な評価に有効です。
RNA品質評価② - 分解度 -
RNAの品質評価には、RNAの分解状態を確認することが重要です。代表的な評価方法としてアガロースゲル電気泳動とBioanalyzerやTapeStationなどの泳動装置があります。アガロースゲルの場合は一定量のRNAが必要なため、微量サンプルでロスを避けたい場合は、そのままお送りいただいても構いません。
分解度について
BioanalyzerやTapeStationは、RNAの分解度を示す指標としてRIN(RNA Integrity Number)を提供します。RINは1から10のスケールで、10が分解のない状態を示し、分解が進むと値が低くなります。
| 図4 RINと分解度合の例 |
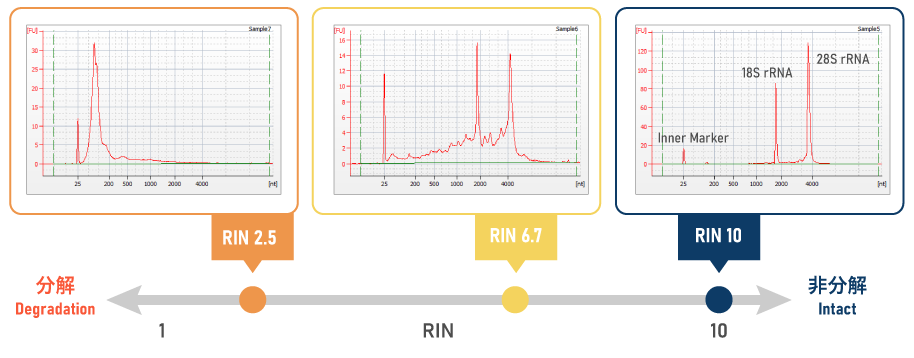 |
分解のないRNAでは、図4の右の泳動像のように、28Sおよび18S rRNAのピークが明瞭で、それ以外の領域にはほとんどRNAが検出されません。分解が進むと、中央の泳動像のように、2つのピークの間や低分子の領域に分解産物が検出され、rRNAのピークが小さくなります。さらに分解が進むと、左の泳動像のようにrRNAのピークが確認できなくなります。
遺伝子発現アレイでは、分解が進んだサンプルを使用すると偽陽性が増加するため、弊社では RIN 7 以上を推奨しています。一方、miRNAアレイではRNAの分解が結果に影響しないため、特に推奨基準はありません。ただし、同一サンプルで遺伝子発現も評価する場合は、分解のない検体を使用することが望ましいです。
哺乳類以外のサンプルについて
RINはHuman、Mouse、Ratなどの典型的なTotal RNAの泳動パターンを基に計算されるため、植物や昆虫など異なるパターンを持つ生物種では正確に計算されない場合があります。この場合は、rRNAのピークが明瞭で他の領域にRNAが少ないことを確認してください。
| 図5 哺乳類以外の泳動像の例 | |
植物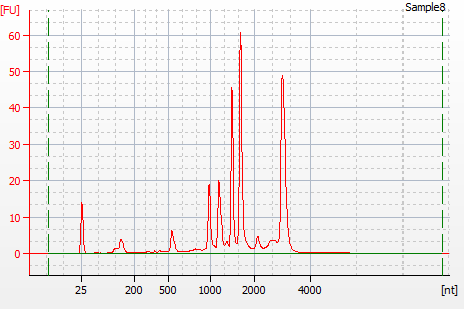 光合成組織ではオルガネラのrRNAが豊富に含まれるため、28S/18S以外にもピークが検出されます。 |
昆虫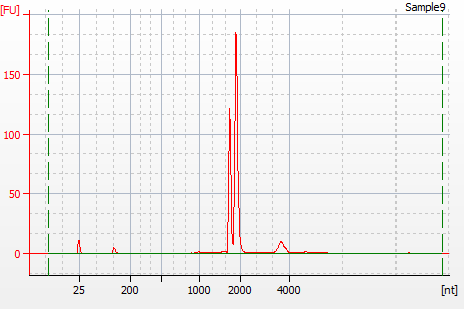 昆虫の28S rRNAは成熟の段階でhidden breakが生じ、18S付近のサイズに切断されています。 |
DNA混入の有無の確認
DNAが混入している場合、図6のようにrRNAとは異なる領域にシグナルが検出されます。このようなサンプルでは、吸光度測定により算出した濃度にDNAが含まれてしまうため、Total RNAの量を正確に測定できない可能性が高くなります。
DNAの混入を確認するには、Qubitなどのターゲット特異的な蛍光光度計での測定が有効です。NanoDropの吸収スペクトルには問題がないのに、NanoDropとQubitの濃度に差がある場合はDNAの混入が疑われます。
DNAの混入はDNase処理によって改善できるため、疑いがある場合は追加精製をお勧めします。
| 図6 DNA混入の例 |
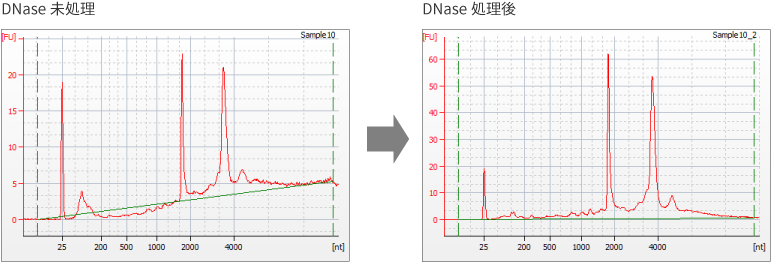 同一サンプルのDNase未処理と、処理後の泳動パターンの変化 同一サンプルのDNase未処理と、処理後の泳動パターンの変化 |