- HOME
- 製品・サービス
- 目的別:アプリケーション選択ガイド
- 微生物ゲノム配列決定
微生物ゲノム配列決定
|
バクテリア専用パッケージプランのご案内
|
||||||||||
解析概要
バクテリアや真菌など、微生物のゲノム配列を決定します。ロングリード解析とショートリード解析、2種類のシーケンス解析手法よりお選びいただけます。
- ロングリード解析
長いコンティグを作成したい場合におすすめです。高分子(>20kb)、かつ、ショートリード解析に比べ大量のゲノムDNAが必要になります。 - ショートリード解析
低コストでドラフトゲノムを作成したい場合や、ゲノムDNAを大量に調製することが難しいサンプルにおすすめです。
解析の流れ
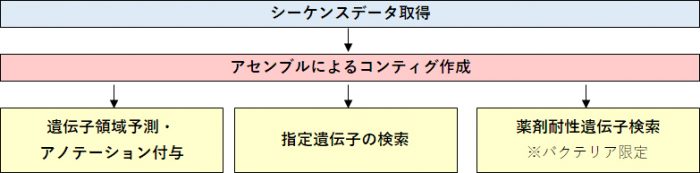
解析メニュー
【De novoアセンブル】
リードのアセンブルを行い、コンティグを作成します。ショートリード解析とロングリード解析のアセンブルの成績についてはQ&Aをご参照ください。
納品データ例:コンティグ配列(FASTA形式)
TTATCAAGAATACTTCGGTTCAATAAAAATGAATCCTGTGGAACTACTCCTAAATTTTGA
TTGATTAATATTTACACCATCAAATAAAATTTTTCCTGTATCAATCTTATATAATCCTGA
…
>scaffold2
AACATTCTCTTCATTTTCAAGCCAGATATCATTTACTCTATTTAAGTATCCTGCTGCCAA
TAAATATTGAGATTTCTGAACTAAATAAAATTGATGCCATGGTTTGAAATGCGACAACCT
…
【Illuminaリードによる塩基補正】
PacBioのリードを使用してアセンブルを行った後、Illuminaのリードを使用してコンティグの塩基補正を行います。
塩基補正の必要性についてはQ&Aをご参照ください。
【遺伝子領域予測・アノテーション】
アセンブルにより得られたコンティグ配列を対象として、ORF 領域を予測します。予測ORF 領域について、既知遺伝子データベースに対する相同遺伝子検索を行い、アノテーション情報を付与します。
納品データ例:アノテーション結果(EXCEL形式)
| ゲノム上の位置情報 | 方向性 | 遺伝子 | 遺伝子産物 | 塩基配列 | アミノ酸配列 |
|---|---|---|---|---|---|
| sequence01:218-1240 | – | pcpC | choline-binding protein F | ATGAAGCTTTTGAAAAA… | MKLLKKMMQVALATFFFG… |
| sequence01:3331-3963 | – | rr03 | DNA-binding response regulator | ATGAAAATTTTACTAGT… | MKILLVDDHEMVRLGLKS… |
| sequence01:3977-4972 | – | hk03 | sensor histidine kinase | ATGAAAAAACAAGCCTA… | MKKQAYVIIALTSFLFVF… |
| sequence01:5744-6754 | – | fni | isopentenyl-diphosphate delta-isomerase | ATGACGACAAATCGTAA… | MTTNRKDEHILYALEQKS… |
| sequence01:6738-7745 | – | mvaK2 | phosphomevalonate kinase | ATGATTGCTGTTAAAAC… | MIAVKTCGKLYWAGEYAI… |
納品データ例:DDBJ登録形式ファイル ※バクテリア限定
| sequence01 | source | 1..278302 | mol_type | genomic DNA |
| organism | Streptococcus pneumoniae | |||
| submitter_seqid | @@[entry]@@ | |||
| ff_definition | @@[organism]@@ @@[strain]@@ DNA, @@[submitter_seqid]@@ | |||
| CDS | complement(218..1240) | product | choline-binding protein F | |
| codon_start | 1 | |||
| inference | COORDINATES:ab initio prediction:MetaGeneAnnotator | |||
| inference | similar to AA sequence:RefSeq:WP_000771073.1 | |||
| gene | pcpC | |||
| locus_tag | LOCUS_00010 | |||
| transl_table | 11 |
【薬剤耐性遺伝子検索】 ※バクテリア限定
バクテリアゲノムを対象として、薬剤耐性遺伝子を検索します。
納品データ例:薬剤耐性遺伝子検索結果(EXCEL形式)
| ゲノム上の位置情報 | 方向性 | 遺伝子名 | 相同性(%) | 薬剤耐性 |
|---|---|---|---|---|
| chromosome1:38381-39151 | + | ant(4′)-Ia | 99.87 | KANAMYCIN/TOBRAMYCIN |
| chromosome1:39368-39772 | + | bleO | 100 | BLEOMYCIN |
| chromosome1:44969-46975 | – | mecA | 100 | METHICILLIN |
| chromosome1:47075-48049 | + | mecR1 | 100 | METHICILLIN |
| chromosome1:126289-127641 | + | tet(38) | 100 | TETRACYCLINE |
| chromosome1:2479070-2479489 | + | fosB-Saur | 99.76 | FOSFOMYCIN |
サンプル必要量
| 解析手法 | ライブラリ調製方法 | 提供サンプル種別 | 総量 | 濃度 | 液量 |
|---|---|---|---|---|---|
| ロングリード解析 | – | 高分子 DNA | 10ug 以上 | 50 ng/uL 以上 | 30 uL 以上 |
| ショートリード解析 | PCR-Plus | DNA | 500 ng 以上 | 10 ng/uL 以上 | 30 uL 以上 |
| PCR-Free | 4 ug 以上 | 50 ng/uL 以上 | 30 uL 以上 |
よくある質問と回答
Q:ロングリードとショートリードのアセンブル結果
ショートリードでは、リピート領域などでアセンブルが難しく、コンティグが分かれて出力されます。ロングリードでは、リピート領域も包含してシーケンスされるため、アセンブルで長いコンティグにまとまります。
アセンブル結果
| ※ 上記は、弊社実績に基づく参考値となります。 |
|||
| 生物種 | ゲノムサイズ | コンティグ数 | |
|---|---|---|---|
| ロングリード解析 | ショートリード解析 | ||
| バクテリア | 1~10Mb | 1~5本 | 30~300本 |
| 真菌 | 10~100Mb | 20~400本 | 200~4,000本 |
Q:バクテリアではロングリード解析によりコンプリートゲノムを作成できますか
PacBioリードを使用したバクテリアゲノムのアセンブル解析では、弊社実績上、多くのサンプルでゲノムが1本のコンティグにまとまります。ただし、サンプルの状態や配列構造により、複数のコンティグに分かれる場合もございます。
Q:PacBioリードのアセンブル後、Illuminaリードを用いた塩基補正は必要ですか
PacBioリードから構築したコンティグ配列には、連続塩基の読み間違い(例:〇 AAAA ⇒ × AAAなど)が見られる傾向があります。このようなInDelは、遺伝子領域予測時のフレームにも影響する可能性があるため、アノテーションを行う前に塩基補正を行うことを推奨しています。
Q:複数に分かれたコンティグをスキャフォールドする方法はありますか
近縁株のコンプリートゲノム配列情報がある場合、そちらをリファレンスにしてコンティグ配列のスキャフォールディングを行うこともできます。ただし、参照株とサンプルのゲノムの構造が異なる領域では誤ったスキャフォールディングが生じる可能性もありますので、コンティグ間の配列についてはキャピラリーシーケンスでもご確認いただくことをお勧めしています。
サービス一覧へ戻る