シーケンサー機種・原理
次世代シーケンサー機種
Illumima
| シーケンサー | RUN条件 | レーンあたりの リード数の目安 |
リード長 | 取得塩基数 | 乗り合い対応 ●:対応可 |
| MiSeq | V2 | 2000万リード (1000万リードペア) |
150 PE | 3Gb | – |
| 250 PE | 5Gb | – | |||
| V3 | 3200万リード (1600万リードペア) |
75 PE | 2.5Gb | – | |
| 300 PE | 10Gb | ● (10万リードペア単位) |
|||
| NextSeq | MidOutput | 2.6億リード (1.3億リードペア) |
75 PE | 19.5Gb | – |
| 150 PE | 39Gb | – | |||
| HighOutput | 4億リード | 75 PE | 30Gb | – | |
| 8億リード (4億リードペア) |
75 PE | 60Gb | – | ||
| 150 PE | 120Gb | – | |||
| NovaSeq | S4 | 52億リード (26億リードペア) |
150 PE | 800Gb | ● (1Gb単位) |
- ※ 上記データ量は参考値であり、保証値ではありません。
PacBio
| シーケンサー | 対応セル | 1ランあたりの リード数の目安 |
リード長 | 取得塩基数 | 乗り合い対応 ●:対応可 |
| Sequel | 1M ZMW | 約50万リード | 10~15kb | 1~3Gb | – |
| Sequel II / IIe | 8M ZMW | 約400万リード | 10~15kb | 15~30Gb | ● ※Sequel II / Revio 機種選択不可 |
| Revio | 25M ZMW | 約1200万リード | 10~15kb | 約50Gb |
- ※ 上記データ量は参考値であり、保証値ではありません。
シーケンス原理
Illumina
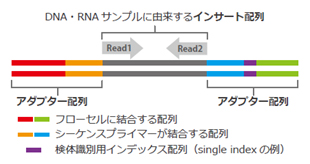
Illuminaのシーケンスライブラリは、DNA・RNAサンプルに由来するインサート配列の両端にアダプターが付いた構造となります。
シーケンサーのフローセルと呼ばれる基板上にライブラリが結合し、1分子のライブラリが増幅してクラスターを形成します。
各クラスターにおいて、1塩基伸長と蛍光読み取りのサイクルを繰り返します。
| <Illuminaシーケンスライブラリ> | <Illuminaシーケンス原理> |
 |
 |
PacBio
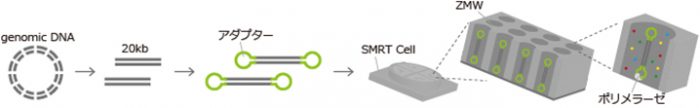
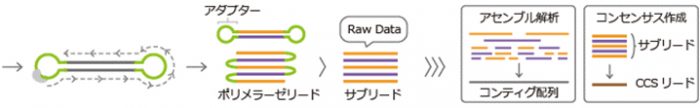
PacBioのシーケンスライブラリは、10~20kbの長いインサート配列の両端に、ヘアピン型のアダプターが付いた構造となります。
SMRT CellにはZMWと呼ばれる小孔があり、1つのZMWで1分子のライブラリのシーケンスが行われます。
1分子のライブラリにおいて、ポリメラーゼが周回しながら数十kbのシーケンスが行われます。
<PacBioシーケンス原理> ※ゲノムシーケンス解析の例
 |
||
| ゲノムDNAを物理的に断片 化し、20kb程度の断片を 回収します。 |
DNA断片の両端に、 ヘアピン型のアダプ ターを付加します。 |
SMRT Cellの孔(ZMW)において、1分子のライブラリと ポリメラーゼが結合し、DNA合成がリアルタイムに検出されます。 |
 |
||
| 環状のライブラリを繰り 返し周回してシーケンス が行われます。 |
出力されるポリメラーゼリード からアダプター配列を除き、サブ リードが得られます。 |
サブリードデータを使用し、以降のデータ解析 を行います。 |
よくある質問と回答
Q:FASTQ形式ファイルとはどのようなデータですか
次世代シーケンサーから出力されるRawデータの形式の一つで、読み取ったリードの「塩基配列」と「クオリティ値」がセットになっている記述様式です。
4行で1本のリードの情報が記述されており、1行目・3行目にリード固有のID、2行目に塩基配列、4行目にクオリティ値が記述されています。
クオリティ値はサンガー法で用いられるPhred Scoreと相関があり、0~42の数値に対応する文字として表記されます。
【 FASTQ形式 】
リードの「塩基配列」と「クオリティ値」の記述様式。
4行で1本のリードの情報が記述され、2行目に塩基配列、4行目にクオリティ値が記載される。
Q:FASTQ形式ファイルを開くことはできますか
FASTQ形式ファイルは、テキストファイルと同様に開くことができます。
ファイルがGZ圧縮されている場合には、解凍を行ってください。
また、ファイルサイズが大きい場合には、巨大テキストファイルに対応した閲覧ソフトをご使用ください。
Q:ペアエンドリードはどのように出力されますか
ペアエンドのRawデータは、Read1とRead2が別々のFASTQ形式ファイルとして出力されます。
[ファイル名]_Read1/Read2.fastq または [ファイル名]_R1/R2.fastq などのように、 同じ接頭名のFASTQファイルにペアのリードが同じ順番で出力されています。
Q:シングルリードとペアエンドはそれぞれどのようなシーケンス方法ですか
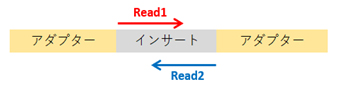
Illuminaのシーケンスではインサート配列の末端からリードの読み取りが行われます。
下図において、シングルリードではRead1のみ、ペアエンドではRead1とRead2がシーケンスされます。
Q:インデックス配列とは何ですか
インデックス配列は、アダプターの中に6~8bp程度含まれる配列で、同じレーンでマルチプレックスシーケンスを行ったサンプルを識別するために使用されます。
弊社のRawデータ出力方法では、リードデータのFASTQファイル内、配列IDの中にインデックス配列が出力されます。
シングルインデックスは片側のアダプター内のインデックスを、デュアルインデックスは両側のアダプター内のインデックスをシーケンスします。
シーケンサーやラン試薬のバージョンにより、インデックスの読み取り方向が異なる場合がございます。

Q:Rawデータにはアダプター配列が含まれていますか
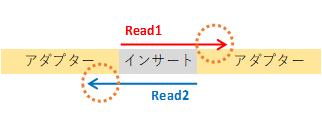
インサートの塩基長がリードの読み取り塩基長よりも短いライブラリでは、リードの3’側にアダプター配列が出現します。
そのため、データ解析を行う前に、3’側のアダプター配列をトリミングしていただくことを推奨しています。
リードの5’側は、アダプター配列の直下からシーケンスが行われるため、基本的にアダプター配列を含みません。
Q:Rawデータについてフィルタリングは行われていますか
弊社のIllumina Rawデータは、基本的にChastityフィルタリング※ を通過したデータを納品させていただいています。
その他のクオリティフィルタリングは行っていません。
正常なシーケンスにおいても、リードの後半では読み取り精度が低下する傾向があるため、必要に応じてクオリティを参照したトリミングやフィルタリングを行ってください。
- ※ Chastityフィルタリング
Illuminaシーケンサーでは、1塩基の伸長反応(1サイクル)ごとにA・T・G・Cの画像データを取得し、個々のリードにおいて、各サイクルで最も強いシグナル値が検出された塩基を採用します。
Chastityフィルタリングでは、以下の計算式に従い、信頼性の低いリードの情報を除外します。
…4種の塩基シグナルの内、最大値をI1、その次に大きい値をI2として、最初の25サイクルまでに{I1/(I1+I2) > 0.6}を満たさないサイクルが2回以上検出されたリードを排除。