- HOME
- 製品・サービス
- 目的別:アプリケーション選択ガイド
- Small RNA-Seq解析
-
核酸原薬CDMO
-
DNA・RNA・修飾核酸
-
次世代シーケンス(NGS)
- 次世代シーケンス(NGS)解析
-
目的別:アプリケーション選択ガイド
- 目的別:アプリケーション選択ガイド
- ヒトゲノム解析
- ゲノムリシーケンス解析(ヒト以外)
- 微生物ゲノム配列決定
- 小スケール解析(NGS Petit)
- GRAS-Di®-ジェノタイピングシーケンス解析
- リピートモチーフ検索
- 一本鎖・微量・損傷 DNA対応ライブラリ調製
- 遺伝子発現解析(リファレンス配列のある生物)
- Iso-Seq 解析(full-length mRNA-seq)
- De novoトランスクリプトーム解析
- Small RNA-Seq解析
- 微生物群集解析
- ショットガンメタゲノム解析
- メタトランスクリプトーム解析
- ChIP-Seq解析
- CRISPRスクリーニング解析
- Illuminaアンプリコンシーケンス解析
- PacBioアンプリコンシーケンス解析
- 標準パイプライン解析
- カスタム解析
- 病理組織標本から遺伝子解析
- シーケンサー機種・原理
- サンプル必要量
- ご注文方法
- NGS論文実績
-
DNAシーケンス
-
マイクロアレイ
-
タンパク質関連
-
関連製品・サービス
Small RNA-Seq解析
解析概要
Small RNA-Seqデータを使用して、miRNAなどのsmall RNAの発現を解析します。
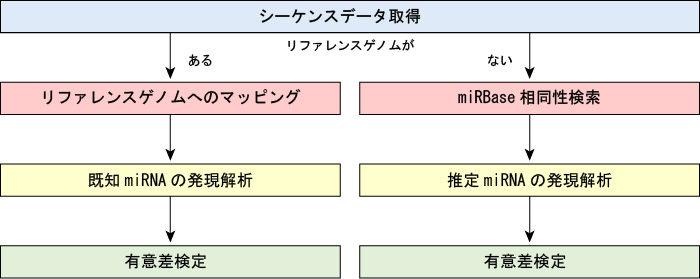
解析の流れ
解析仕様
| 使用機器 | Illumina NovaSeq |
| データ量 | 1,000万リードペア/検体 |
解析メニュー
【リファレンスゲノムマッピング・既知miRNAの発現解析】
small RNA-Seqのリードをリファレンスゲノムにマッピングし、既知miRNAの発現頻度を算出します。
標準解析では、検体間・群間のFold Change算出まで実施します。
【miRbase相同性検索・推定miRNAの発現解析】
small RNA-Seqのリードの同一配列を集計し、既知miRNAとの相同配列検索を行います。
得られた推定miRNAについて、リードカウント情報を基に発現頻度を予想し、検体間・群間比較を行いFold Changeを算出します。
【有意差検定】
群間におけるmiRNA発現変動の有意差検定を行います。(n≧3 推奨)
サンプル必要量
| 提供サンプル種別 | 総量 | 濃度 | 液量 |
| total RNA | 2 ug 以上 | 50 ng/uL 以上 | 30 uL 以上 |
| Small RNA | 60 ng 以上 | 2 ng/uL 以上 | 30 uL 以上 |