- HOME
- 製品・サービス
- 目的別:アプリケーション選択ガイド
- ヒトゲノム解析
-
核酸原薬CDMO
-
DNA・RNA・修飾核酸
-
次世代シーケンス(NGS)
- 次世代シーケンス(NGS)解析
-
目的別:アプリケーション選択ガイド
- 目的別:アプリケーション選択ガイド
- ヒトゲノム解析
- ゲノムリシーケンス解析(ヒト以外)
- 微生物ゲノム配列決定
- 小スケール解析(NGS Petit)
- GRAS-Di®-ジェノタイピングシーケンス解析
- リピートモチーフ検索
- 一本鎖・微量・損傷 DNA対応ライブラリ調製
- 遺伝子発現解析(リファレンス配列のある生物)
- Iso-Seq 解析(full-length mRNA-seq)
- De novoトランスクリプトーム解析
- Small RNA-Seq解析
- 微生物群集解析
- ショットガンメタゲノム解析
- メタトランスクリプトーム解析
- ChIP-Seq解析
- CRISPRスクリーニング解析
- Illuminaアンプリコンシーケンス解析
- PacBioアンプリコンシーケンス解析
- 標準パイプライン解析
- カスタム解析
- 病理組織標本から遺伝子解析
- シーケンサー機種・原理
- サンプル必要量
- ご注文方法
- NGS論文実績
-
DNAシーケンス
-
マイクロアレイ
-
タンパク質関連
-
関連製品・サービス
ヒトゲノム解析
解析概要
ヒトのゲノムシーケンス解析により、疾患等の原因となる変異候補箇所を検索します。
解析手法
ホールゲノムシーケンス解析では、イントロンや遺伝子間領域も解析対象とすることができますが、カバレッジを深く読むことが難しいため、主にgermline mutationの検出に用いられます。エクソームシーケンス解析では、高いカバレッジにより、がん組織などで低頻度の変異を検出することができます。
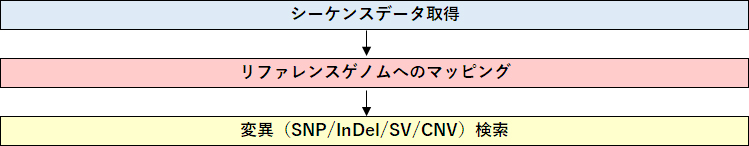
解析の流れ
推奨データ取得量
| 解析手法 | ターゲット領域 | ターゲットサイズ | 解析例 | |
| 取得データ量 | カバレッジ※2 | |||
| ホールゲノムシーケンス | 全ゲノム | 3Gb | 90Gb | ×30 |
| エクソームシーケンス※1 | 全エクソン | 60Mb | 6Gb | ×50 |
- ※1 Agilent SureSelect Human All Exon V6
- ※2 エクソームシーケンスのカバレッジは、キャプチャ効率やバイアスを加味した参考値を記載しています。
解析メニュー
【マッピング・変異候補検索】
リファレンスゲノムにリードをマッピングし、SNP・InDel・SV・CNVの変異候補箇所を検索します。
- ※ SV・CNVはホールゲノムシーケンスでのみ解析可能です。
サンプル必要量
| 解析手法 | ターゲット領域 | ライブラリ調製方法 | 提供サンプル種別 | 総量 | 濃度 | 液量 |
| ホールゲノムシーケンス | 全ゲノム | PCR-Plus | DNA | 500 ng 以上 | 10 ng/uL 以上 | 30 uL 以上 |
| PCR-Free | 4 ug 以上 | 50 ng/uL 以上 | 30 uL 以上 | |||
| エクソームシーケンス | 全エクソン | PCR-Plus | 2 ug 以上 | 10 ng/uL 以上 | 30 uL 以上 |
よくある質問と回答
Q:カバレッジと変異の頻度
カバレッジは、ターゲット領域中の任意のポジションをカバーするリードの平均本数に相当し、「取得データ量(bp)÷ ターゲットサイズ(bp)」で算出されます。また、エクソーム解析では、キャプチャーの効率やバイアス等により、上記で計算した値よりも実際の平均カバレッジは減少します。
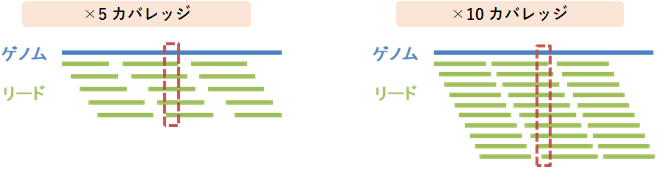
変異の頻度とは、サンプル中の細胞の中で、任意の変異を有する細胞の割合です。
例えば、サンプルの中に4割存在するがん細胞で共通して有するヘテロの変異は、変異頻度20%となります。
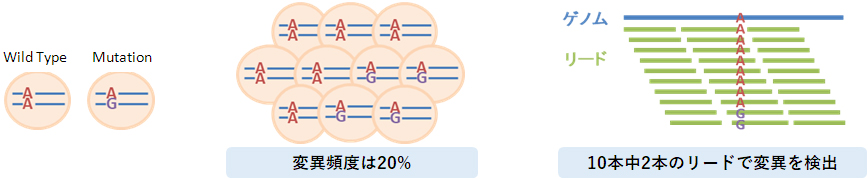
Q:コントロールサンプルは必要ですか
疾患の有無に関わらず、1検体のみで変異検索を行うと、リファレンスゲノムとサンプルのゲノムとの個体差や、ミスマッピングに由来する変異候補も多数検出されてしまいます。(ヒトホールゲノム解析では400万箇所、エクソーム解析では50万箇所程度)
コントロールサンプルとして、somatic mutationの場合は同一検体の正常組織サンプル、germline mutationの場合は近縁な検体が使用されます。コントロールサンプルで検出される変異候補箇所をバックグラウンドとすることで、対象サンプルで特異的な変異を抽出することができます。
Q:低頻度変異が検出された場合、バリデーションを取る方法はありますか
変異箇所領域のPCR を行い、PCR 産物のディープシーケンスにより変異の存在と頻度を確認することができます。(アンプリコンシーケンス解析参照)