- HOME
- 製品・サービス
- 目的別:アプリケーション選択ガイド
- ChIP-Seq解析
-
核酸原薬CDMO
-
DNA・RNA・修飾核酸
-
次世代シーケンス(NGS)
- 次世代シーケンス(NGS)解析
-
目的別:アプリケーション選択ガイド
- 目的別:アプリケーション選択ガイド
- ヒトゲノム解析
- ゲノムリシーケンス解析(ヒト以外)
- 微生物ゲノム配列決定
- 小スケール解析(NGS Petit)
- GRAS-Di®-ジェノタイピングシーケンス解析
- リピートモチーフ検索
- 一本鎖・微量・損傷 DNA対応ライブラリ調製
- 遺伝子発現解析(リファレンス配列のある生物)
- Iso-Seq 解析(full-length mRNA-seq)
- De novoトランスクリプトーム解析
- Small RNA-Seq解析
- 微生物群集解析
- ショットガンメタゲノム解析
- メタトランスクリプトーム解析
- ChIP-Seq解析
- CRISPRスクリーニング解析
- Illuminaアンプリコンシーケンス解析
- PacBioアンプリコンシーケンス解析
- 標準パイプライン解析
- カスタム解析
- 病理組織標本から遺伝子解析
- シーケンサー機種・原理
- サンプル必要量
- ご注文方法
- NGS論文実績
-
DNAシーケンス
-
マイクロアレイ
-
タンパク質関連
-
関連製品・サービス
ChIP-Seq解析
解析概要
ChIP-Seqでは、ゲノム上の転写因子結合部位やヒストン修飾領域を網羅的に検出することができます。
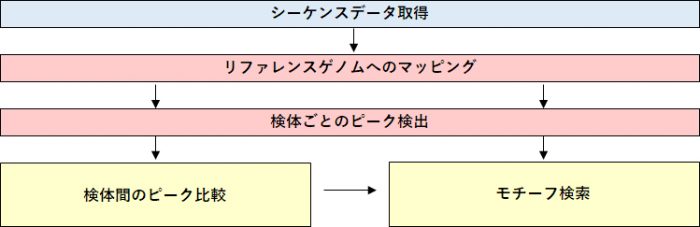
解析の流れ
推奨データ取得量
| シーケンス仕様 | llumina 150PE |
| データ量 | 2,000万リードペア/検体 |
解析メニュー
【マッピング・ピーク検出】
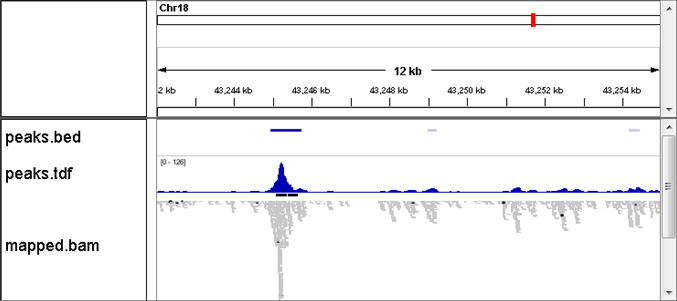
リードをリファレンスゲノムにマッピングし、ピーク領域を検出します。
マッピング結果
【検体間のピーク領域比較】
複数検体のピーク領域について、比較テーブルを作成します。検体間で共通したピークや、一方のサンプルで特異的なピークを抽出することができます。
【モチーフ検索】
ゲノムワイドに得られたピーク領域の塩基配列を対象として、高頻度で出現する配列モチーフを検索します。
納品データ例:シーケンスロゴ

サンプル必要量
| 提供サンプル種別 | 総量 | 濃度 | 液量 |
| ChIP-DNA | 20 ng 以上 | 1 ng/uL 以上 | 30 uL 以上 |
よくある質問と回答
Q:シングルリードのシーケンスはできますか
弊社では、Illumina 150bp ペアエンドシーケンスの乗り合い解析を行っていますので、基本的にはペアエンドシーケンスをご案内させていただきます。
- ※ ペアエンドのRead1がシングルリードに相当します。
Q:コントロールサンプルは必要ですか
ChIP-Seqでは、バックグラウンドのノイズを除去するために、input DNAやIgG-ChIP DNAなどのコントロールサンプルをご用意いただくことを推奨いたします。
〇 input DNAサンプル
input DNAは、IP処理を行わずに断片化したゲノムDNAのサンプルです。DNA断片化やPCR増幅で生じるバイアスに由来したピークが得られ、バックグラウンドデータとして使用することができます。
〇 IgG ChIPサンプル
IgGのChIPを行ったサンプルでは、IgGへの非特異的な結合の情報が得られます。非特異的結合が少ない場合には、十分なDNA量を回収することができず、次世代シーケンス解析を行うことが困難になりますので、その場合には上記InputDNAをコントロールとしてご提供ください。