GEO登録サポートページ
GEOとは
GEO(Gene Expression Omnibus)は、NCBIが運営する公共のデータベースで、遺伝子発現データを収集、保存、共有するためのプラットフォームです。GEOは主にマイクロアレイ、次世代シーケンス、その他の高スループットの遺伝子発現解析技術から得られたデータを扱っています。
マイクロアレイなどの遺伝子発現解析を含む論文を投稿する際に、GEOへのデータ登録を求められる場合があります。当ページにGEO登録に関する情報をまとめました。
用語解説
- Metadata(メタデータ)
- メタデータは、実験の背景情報をまとめたファイルです。タイトル、サマリー、実験条件、サンプルの詳細、使用したプラットフォームなど、データを理解するために必要な付随情報を含みます。
- Matrix Table(マトリックステーブル)
- マトリックステーブルは、サンプルごとの遺伝子発現値を表形式に表したデータです。行には遺伝子、列にはサンプルが配置され、それぞれの交差点に発現値が記載されています。
- Platform(プラットフォーム)
- プラットフォームは、遺伝子発現データを取得するために使用された技術や機器のことを指します。GEOでは、マイクロアレイチップ(スライド)やシーケンシング技術ごとにデータが分類されており、各プラットフォームには “GPL” で始まるIDが付けられています。
- Raw Data(ローデータ/生データ)
- 生データは、実験から直接得られた未処理のデータです。アジレントアレイは数値化ソフトから得られたtxtファイル、次世代シーケンスではfastqファイルを指します。アジレントアレイのRawデータについて詳しくはこちらもご参考ください。
- Processed Data(プロセスドデータ/解析データ)
- 解析データは、生データに対してデータQC、ノーマライズや補正などの処理を施した後のデータです。最終的な発現値テーブルや解析結果が含まれます。
- Sample(サンプル)
- GEOでは各サンプルに対して、条件、処理内容、取得方法などの詳細が記載されます。サンプルごとに “GSM” で始まるIDが付与されます。
- Series(シリーズ)
- GEOではひとつの実験をシリーズと呼び、実験概要や目的がまとめられています。シリーズごとに “GSE” で始まるIDが付与されます。論文に記載するaccessionは、基本的にこのGSEのIDになります。同じサンプルの遺伝子発現データとmiRNA発現データのように、複数のシリーズを関連付けてスーパーシリーズ(SuperSeries)として登録する場合もあります。
- Submit(サブミット)
- Submitは、GEOへ研究者が遺伝子発現データを提出(フォーム送信 や アップロード)するプロセスを指します。提出データはメタデータ、Rawデータ、Processedデータで構成されます。
- Release Date(リリースデイト/公開日)
- GEOでは、データ公開日を設定することができます。公開日はデータ提出後も任意に変更することができます。最大4年先まで指定可能ですが、論文もしくはプレプリントが公開されたらGEOデータも公開する必要があります。
GEO登録の流れ
-
STEP 1アカウント作成GEOにデータを提出するには、NCBIアカウントと、それに付随するMy GEO Profileの登録が必要です。
① NCBIアカウント作成ページからアカウントを作成してください。
② NCBIアカウントにログインした状態でMy GEO Profileに所属、名前、連絡先などの情報を入力します。 -
STEP 2データの準備GEOからテンプレートファイルをダウンロードし、サンプル情報、実験条件、プラットフォーム情報など、背景情報を記載したメタデータを作成します。
提出にはメタデータ、Rawデータ、Processedデータが揃っている必要があります。 -
STEP 3データの提出GEO Submission Web form または FTP を使用してデータを送信します。
提出時にデータ公開日を設定します。(あとから変更することもできます) -
STEP 4登録完了NCBIのスタッフがデータを検証し、不備がないかを確認します。
検証が完了すると、登録のお知らせがメールで届き accession が付与されます。
Metadata記載内容について
Protocol記載例
ここでは、GEO の Metadata 記載例の英文を基に、2024年現在の北海道システム・サイエンスで最もよく使用される実験条件で改変した例文を載せています。
内容は登録データやご注文内容、ご依頼時期によって変わりますので、実際に使用された条件については納品データをご確認ください。また、必ずこの文章を使用しなければならないというものではなく、書き方はご自身で適宜変更ください。
ピンクのマーカー部分は実験に応じて内容が変わる可能性があります。
遺伝子発現アレイ 1カラー
| label protocol | Cyanine-3 (Cy3) labeled cRNA was prepared from 50 ng Total RNA using Low Input Quick Amp Labeling Kit (Agilent) according to the manufacturer’s instructions, followed by RNAeasy column purification (QIAGEN). Dye incorporation and cRNA yield were checked with the NanoDrop ND-1000 Spectrophotometer. |
| hyb protocol | 600 ng of Cy3-labelled cRNA (specific activity >6 pmol Cy3/ug cRNA) was fragmented at 60°C for 30 minutes in a reaction volume of 25 ul containing 25x Agilent fragmentation buffer and 10x Agilent blocking agent following the manufacturers instructions. On completion of the fragmentation reaction, 25 ul of 2x Agilent GE hybridization buffer HI-RPM was added to the fragmentation mixture and hybridized to SurePrint G3 Human GE 8x60K Ver.3.0 Microarray (Agilent, G4851C) for 17 hours at 65°C in a rotating Agilent hybridization oven. After hybridization, microarrays were washed 1 minute at room temperature with GE Wash Buffer 1 (Agilent) and 1 minute with 37°C GE Wash buffer 2 (Agilent). |
| scan protocol | Slides were scanned immediately after washing on the Agilent DNA Microarray Scanner (G2600D) using one color scan setting for 8x60k array slides (Scan Area 61×21.6 mm, Scan resolution 3um, Dye channel is set to Green and Green PMT is set to 100%). |
| data processing | The scanned images were analyzed with Feature Extraction Software 12.0.3.1 (Agilent) using default parameters (protocol GE1_1200_Jun14 and Grid 072363_D_F_20200924) to obtain background subtracted and spatially detrended Processed Signal intensities. The 75th percentile shift normalization was performed using Agilent GeneSpring GX 14.9.1. |
| value definition | Normalized signal intensity |
miRNAアレイ
| label protocol | RNA labeling was performed using miRNA Complete Labeling Reagent and Hyb Kit (Agilent, 5190-0456) following the Agilent protocols v.2.4. 100 ng of total RNA was dephosphorylated with calf intestine alkaline phosphatase for 30 min at 37°C. After adding DMSO, the reaction mixture was incubated at 100°C for 7 minutes and immediately transferred to ice water bath. Ligation was performed with Cy3-pCp at 16 °C for 2 hours.The labeled RNA were dried completely in a vacuum concentrator and resuspended in 18 ul of nuclease free water. |
| hyb protocol | The hybridization mixture was denatured at 100°C for 5 minutes in a reaction volume of 45 ul containing 2x Agilent GE hybridization buffer HI-RPM and 10x Agilent blocking agent following the manufacturers instructions and immediately transferred to ice water bath. Each sample was hybridized to SurePrint G3 Human miRNA microarray 8 x 60K Rel.21.0 (Agilent, G4872A#70156) for 20 hours at 55°C in a rotating Agilent hybridization oven. After hybridization, microarrays were washed 1 minute at room temperature with GE Wash Buffer 1 (Agilent) and 5 minute with 37°C GE Wash buffer 2 (Agilent). |
| scan protocol | Slides were scanned immediately after washing on the Agilent DNA Microarray Scanner (G2600D) using miRNA setting for 8x60k array slides (Scan Area 61×21.6 mm, Scan resolution 3um, Dye channel is set to Green and Green PMT is set to 100%). |
| data processing | The scanned images were analyzed with Feature Extraction Software 12.0.3.1 (Agilent) using default parameters (protocol miRNA_1200_Jun14 and Grid 070156_D_F_20141006) to obtain background subtracted and spatially detrended Processed Signal intensities. The 90th percentile shift normalization was performed using Agilent GeneSpring GX 14.9.1. |
| value definition | Normalized signal intensity |
| ・使用したスライド名 | … | 作業報告書(pdf) |
| ・実験の詳細条件 | … | [protocol] フォルダ内のpdfファイル |
| ・数値化プロトコルとグリッド | … | Rawデータtxtファイルをエクセルで開き、B3セルとJ3セル |
Platformの探し方
アジレントアレイの場合、スライドの種類ごとにデザインIDという5桁(もしくは頭に0を付けた6桁)の番号がアジレント社より設定されています。このデザインIDを使って、該当するGEOプラットフォームを探すのが最も分かりやすいです。
弊社納品データのRawデータは下記のようなファイル名になっています。
SG12345678_257236300001_S001_GE1_1200_Jun14_1_1.txt
左から1番目と2番目の”_”に挟まれた12桁の番号がスライド番号を示しており、特に赤線部分(頭の25を除く次の5桁)の「72363」がデザインIDを示しています。
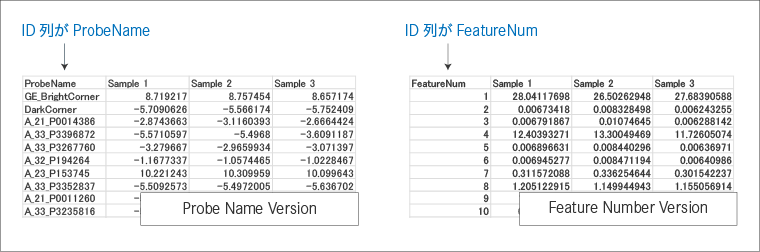
この「72363」を GEO Platform 一覧ページの検索ボックスに入力すると検索結果がいくつか出てきますが、一般的には”Contact”が”Agilent Technologies”になっている [Probe Name Version] か [Feature Number Version] のケースがほとんどです。どちらのバージョンかは、登録データが「ProbeNameをIDとする発現テーブル」になっているか、「FeatureNumをIDとする発現テーブル」になっているかで判断します。
お問い合わせ
昔のデータで実験条件が分からない等のお悩みがございましたら、可能な限りお調べしますので下記よりお問い合わせください。「受注番号」や「当時のご依頼者名」がもしお分かりでしたら、ご教示いただけますとスムーズに調査ができます。
手順に不安がある方や手間を省きたい方には、有料で登録代行作業も承ります。ぜひご相談ください。
サービス一覧へ戻る